クロマチン構造の基本的単位 ヌクレオソーム (nucleosome )は、真核生物 におけるDNA のパッケージングの基本的単位であり、クロマチン の基本要素である。ヌクレオソームは、ヒストン八量体 (histone octamer ) とその周囲に巻きついたDNAから構成される[ 1] H2A 、H2B 、H3 、H4 各2コピーずつから成る。
より正確には、ヌクレオソームは、ヌクレオソームコア粒子 (nucleosome core particle) とリンカーDNA (linker DNA) から構成される。ヌクレオソームコア粒子とは、ヒストン八量体の周囲を約146 bpのDNAが左巻きに1.67周分巻いて形成される構造であり[ 2] [ 3]
ヌクレオソーム構造は、1974年にDon OlinsとAda Olinsによって電子顕微鏡 を用いて初めて観察された[ 4] ロジャー・コーンバーグ (Roger Kornberg ) によって提唱された[ 5] [ 6] in vitro で[ 7] マイケル・グルンスタイン (Michael Grunstein ) によってin vivo で実証された[ 8]
ヌクレオソームは、コアヒストンの翻訳後修飾 という形でエピジェネティック な遺伝情報を運ぶと考えられている。ゲノム 中のヌクレオソームの位置はランダムではなく、各ヌクレオソームの位置によって調節タンパク質のDNAへのアクセシビリティが決定される[ 9]
真核生物のクロマチンは、ヌクレオソーム形成を基盤として、より複雑な構造へと折りたたまれ、細胞核 内に収納される[ 10] [ 11] [ 12]
ほとんどの真核細胞とは対照的に、成熟した精細胞 はゲノムDNAのパッケージングに主にプロタミン を利用する。これは、はるかに高いパッケージングを達成するためである可能性が高い[ 13]
古細菌 でもヒストンに対応するものや単純なクロマチン構造が発見されており[ 14]
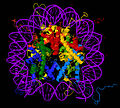
H2A 、H2B 、H3 、H4 コアヒストンとDNAから構成されるヌクレオソームコア粒子。超らせん軸方向から。
1980年代のAaron Klugのグループによる先駆的な構造研究により、ヒストンタンパク質八量体の周囲にDNAが左巻き超らせんで約1.7ターン巻き付いているという最初の証拠が得られた[ 15] 結晶構造 がRichmondのグループによって解かれ、粒子の最も重要な詳細が示された。この結晶構造を得るのに重要であったヒトαサテライト の回文配列 DNAは、オークリッジ国立研究所 のBunickのグループによって開発されたものである[ 16] [ 17] [ 18] [ 19] [ 20] [ 21] ヒストンバリアント を含むものや異なる生物種のヒストンも含むものも含まれている。ヌクレオソームコア粒子の構造は顕著に保存されており、カエルと酵母のヒストンは100残基以上が異なるが、全体での二乗平均平方根誤差 (RMSD)はわずか1.6 Åである[ 22]
ヌクレオソームコア粒子には約146 bpのDNAが含まれ、ヒストンH2A、H2B、H3、H4各2コピーずつからなるヒストン八量体の周囲に左巻き超らせんで約1.67周分巻き付いている。隣接するヌクレオソームとは、リンカーDNAと呼ばれる拘束されていないDNAによって連結されている。リンカーDNAの長さは生物種や組織によって、10–80 bp程度と差がある[ 14]
アポトーシスによるDNA断片化 (英語版 ) ヌクレオソームの構成の模式図[ 23] ヌクレオソームコア粒子の結晶構造PDB : 1EQZ [ 24] [ 25] ヌクレオソームコア粒子は、間期 のクロマチンを部分的にほどく処理を行った際に観察される。電子顕微鏡によって得られた像は、ひもでつながったビーズ(beads-on-a-string)のような形状をしており、ひもがDNA、ビーズ1つ1つがヌクレオソームコア粒子である。ヌクレオソームコア粒子はDNAとヒストンタンパク質から構成される[ 26]
DNase によるクロマチン の部分分解によって、ヌクレオソームの構造は明らかにされる。リンカー部分と比較してヌクレオソームコア粒子のDNAにはDNaseがアクセスしにくいため、DNAはヌクレオソーム間の距離の倍数に等しい長さの断片(180 bp、360 bp、540 bp、…)へと分解される。したがって、部分分解を行ったDNAのゲル電気泳動では、はしご(ラダー)状の非常に特徴的なパターンが観察される[ 23] アポトーシス (プログラム細胞死)時など自然条件下でも生じる。
コアヒストンタンパク質はヒストンフォールド (英語版 ) αヘリックス (α1–3)から構成される。溶液中では、ヒストンタンパク質はH2A-H2Bヘテロ二量体とH3-H4ヘテロ四量体を形成する。ヒストンは長いα2ヘリックスを介して逆平行方向に二量体化する。さらに2つのH3-H4二量体は、H3どうしの広範囲にわたる相互作用によって安定化された4ヘリックスバンドル を形成する。H2A-H2B二量体は、H4-H2B間の相互作用によってH3-H4四量体に結合する。H4-H2B間の相互作用には疎水的クラスターの形成が含まれる[ 2]
ヌクレオソームには120か所以上の直接的なタンパク質-DNA間相互作用と、数百か所の水分子を介した相互作用が含まれている[ 27] ヒドロキシル基 、主鎖のアミド がDNA骨格のリン酸 基と塩橋 や水素結合 を形成し、DNAとの相互作用の大部分を担う。ヌクレオソームがゲノム上に遍在的に分布することを考えると、それが配列非特異的なDNA結合因子であることは重要である。ヌクレオソームは一部のDNA配列に対して選択性を示す傾向があるが[ 28] デオキシリボース の間では非極性相互作用が形成され、DNAの副溝が八量体表面に面している14か所すべてでアルギニン 側鎖がインターカレート している。DNA結合部位の分布と結合強度はヌクレオソームコア内のDNAを変形させる。DNAの屈曲は一様ではなく、ねじれ欠陥(twist defect)を有する。溶液中のB型DNAではねじれは1ターンあたり10.5 bpである。しかし、ヌクレオソーム内のDNAの全体的なねじれは1ターンあたり10.2 bpであり、その値は9.4から10.9まで変動する。
ヒストンテールはヒストンの質量の最大30%を占るが、高度な柔軟性のためヌクレオソームの結晶構造では可視化されず、大部分は安定な構造を形成しないと考えられている[ 29] アセチル化 はクロマチンの高次構造を歪めることが示唆される。
現在のクロマチンの凝縮のモデル ヒストンH1 とそのアイソフォーム などのリンカーヒストンはクロマチンのコンパクト化に関与しており、これらはヌクレオソームのDNAの入口と出口の近傍に位置してリンカー領域のDNAと結合している[ 30] [ 31]
テトラヌクレオソーム(4ヌクレオソーム)の結晶構造が解かれ、それをもとに30 nm繊維の二条らせん(two-start helix)構造が提唱された[ 32] 電子顕微鏡 データとは相容れず、いまだに一定の論争がある[ 33] ユークロマチン を形成すると考えられている。さらに圧縮されると、転写不活性なヘテロクロマチン となる。
ヌクレオソームは非常に安定なタンパク質-DNA複合体であるが、静的な複合体ではなく、ヌクレオソームスライディング(nucleosome sliding)やDNA の自発的露出など、さまざまな構造的再構成が行われることが示されている。状況に依存して、ヌクレオソームは転写因子の結合を阻害したり促進したりする。ヌクレオソームの配置には3つの主要な要素の寄与によって制御されている。1つ目に、ヒストン八量体の結合親和性はDNA配列に依存する。2つ目に、ヌクレオソームは他のタンパク質因子の競合的結合によって除去されたり、協働的結合によってリクルートされたりする。3つ目に、ヌクレオソームはATP依存性リモデリング複合体によって移動される場合がある[ 34]
Bradburyの研究室によって、5S DNA上に再構成されたヌクレオソームは熱を加えることで近接配列へ移動して再配置されることが示された[ 35] シス に「スライド」することが示された。さらに2008年には、CTCF 結合部位がヌクレオソームの配置のアンカー部位として機能することが明らかにされ、さまざまなゲノムシグナルの解析の際のアラインメントに利用することで、隣接して位置する複数のヌクレオソームに関する情報を容易に得られるようになった[ 36] クロマチンリモデリング 酵素の大きなファミリーを進化させており、その多くはヌクレオソームスライディングを利用する。2012年、Beena Pillaiの研究室はヌクレオソームスライディングが大規模な組織特異的な遺伝子発現の機構の1つである可能性を示した。この研究では、特定の組織で発現している遺伝子の転写開始部位にはヌクレオソームが存在しないが、その遺伝子が発現していない他の組織ではヌクレオソームが結合していることが示された[ 12]
Widomの研究室の研究によって、ヌクレオソームのDNAは巻き付いた状態と巻き付いていない状態の平衡にあることが示された。時間分解FRET を用いたその速度の測定によって、ヌクレオソーム内のDNAは約250 msの間完全に巻き付いた状態が維持された後に自発的にほどけ、10–50 ms以内に再び巻き付くことが明らかにされた[ 37] [ 38] RNAポリメラーゼII の前進の制限に重要な役割を果たしている[ 39]
活発な遺伝子のプロモーター には、ヌクレオソームを含まない領域(nucleosome free region、NFR)が存在する。それによってプロモーターDNAに転写因子などさまざまなタンパク質がアクセスできるようになる。一般的に出芽酵母 Saccharomyces cereviae では、NFRは約200ヌクレオチドにわたって存在している。安定した位置にあるヌクレオソーム(well-positioned nucleosome)がNFRの境界を形成する。こうしたヌクレオソームは+1-ヌクレオソーム、−1-ヌクレオソームと呼ばれており、転写開始部位からそれぞれ下流、上流の一定の距離に位置している。+1-ヌクレオソームとその下流のいくつかのヌクレオソームにはヒストンバリアントH2A.Z (英語版 ) [ 40]
真核生物のゲノムはクロマチンを形成しているが、細胞は大量のクロマチンとは無関係に特定の遺伝子座 を空間的・時間的に調節する必要がある。DNA複製、修復、転写などの核内過程の調整に必要な高いレベルの制御を達成するため、細胞はクロマチンの構造と機能を局所的・特異的に調節するさまざま手段を発達させている。これには、ヒストンの共有結合修飾、ヒストンバリアントの組み込み、ATP依存的リモデリング酵素による非共有結合的な再構成などがある。
ヒストンテールと、クロマチン形成におけるその機能 ヒストン修飾は1960年代半ばに発見されて以降、転写に影響を与えることが予測されてきた[ 41] [ 42] [ 43] [ 44] アセチル化 やリン酸化 など、ヒストンコアの電荷を減らす修飾はコアとDNAの結合を緩めることが予測される。その影響の大きさはコア内の修飾の位置に依存している[ 45] サイレンシング と相関しており、他のものは遺伝子の活性化と相関しているようである。一般的な修飾としては、リジン のアセチル化、メチル化 、ユビキチン化 、アルギニン のメチル化、セリン のリン酸化である。このようにして保存された情報は、DNAにはコードされていないものの娘細胞に遺伝するため、エピジェネティック な遺伝とみなされる。遺伝子の抑制状態や活性化状態の維持は、細胞分化 の過程で必要となることが多い[ 14]
ヒストンは進化の過程で顕著に保存されているが、いくつかのバリアントが同定されている。このヒストンの機能の多様化はH2AとH3にほぼ限られており、H2BとH4はほぼ不変である。H2AはH2A.Z(ヌクレオソームの安定性の低下をもたらす)またはH2A.X (DNA修復 やT細胞 の分化と関係する)によって置き換えられる場合があり、哺乳類の不活性化X染色体 にはmacroH2A (英語版 ) セントロメア ではH3はCENPA によって置き換えられる[ 14]
多数の異なる反応がATP 依存性クロマチンリモデリング という用語で呼ばれている。リモデリング酵素は、ヌクレオソームをDNAに沿ってスライドさせたり[ 46] [ 47] [ 48] 超らせん ねじれを形成したりする[ 49] [ 50]
In vivo での遺伝子活性化[ 51] in vitro でのリモデリング[ 52] 胚性幹細胞 の発生過程でヌクレオソームの位置は大きく変化し、こうした変化は発生に関係した転写因子の結合と関係していることが示されている[ 53]
2007年の研究では、酵母ゲノム中のヌクレオソームの位置の一覧が作成され、プロモーター 領域や複製起点 ではヌクレオソームが失われていることが示された[ 54] [ 55] [ 56] [ 57] [ 57] [ 58] 出芽酵母 Saccharomyces cerevisiae のゲノム全体でのヌクレオソームの位置変化が明らかにされた[ 59] [ 60] [ 61]
In vitro でのヌクレオソームの組み立てヌクレオソームの組み立ての模式図 ヌクレオソームは、精製された天然のヒストンまたは組換えヒストンのいずれかを用いてin vitro [ 62] [ 63] 透析 を用いるものである。ヒストン八量体と裸のDNAを2 M の塩濃度の反応溶液で共にインキュベートし、塩濃度を徐々に低下させることで、DNAはヒストン八量体の周囲に巻きついた状態に平衡化され、ヌクレオソームが形成される。適切な条件下でこの再構成過程を行うことによって、特定の配列のヌクレオソーム配置親和性の実験的マッピングを行うことができる[ 64]
近年、部位特異的ジスルフィド 架橋によって安定性が向上したヌクレオソームコア粒子を生産することが可能となっている[ 65] [ 66]
In vivo でのヌクレオソームの組み立てヌクレオソームの組み立てのステップ ヌクレオソームは周囲にDNAが巻き付いたヒストンタンパク質から構築される、DNAのパッキングの基本的単位である。ヌクレオソームはより高次のクロマチン構造の形成のための足場となるとともに、遺伝子発現制御の1つの階層を形成している。DNA複製 の過程で解体されたヌクレオソームは、複製フォークの後ろで、新たに合成されたDNA上に迅速に組み立てられる。
古いヌクレオソームから解体されたヒストンH3とH4は近接して存在し続け、新たに合成されたDNA上にランダムに分布する[ 67] CAF-1 (英語版 ) [ 68] ASF1 (英語版 ) [ 69] [ 70] ヒストン修飾酵素 をリクルートして新しいヒストンに標識を付加することで、エピジェネティックな記憶に寄与する。
古いH3やH4の場合とは対照的に、古いH2AとH2Bは放出されて分解される。そのため、新たなヌクレオソームには新たに組み立てられたH2AとH2Bが組み込まれる[ 71] [ 72] Ino80 やChd1 (英語版 ) [ 73] [ 74]
ヌクレオソームコア粒子の結晶構造(PDB : 1EQZ [ 24] [ 25] H2A 、H2B 、H3 、H4 、DNA と色付けされている。
^ Reece, Jane; Campbell, Neil (2006). Biology ISBN 978-0-8053-6624-2 . https://archive.org/details/biologyc00camp ^ a b “Crystal structure of the nucleosome core particle at 2.8 A resolution”. Nature 389 (6648): 251–60. (September 1997). Bibcode : 1997Natur.389..251L . doi :10.1038/38444 . PMID 9305837 .
^ Alberts, Bruce (2007). Molecular biology of the cell (5th ed.). New York: Garland Science. p. 211. ISBN 978-0-8153-4106-2 ^ “Spheroid chromatin units (v bodies)”. Science 183 (4122): 330–2. (January 1974). Bibcode : 1974Sci...183..330O . doi :10.1126/science.183.4122.330 . PMID 4128918 . ^ “Milestone 9, (1973-1974) The nucleosome hypothesis: An alternative string theory” . Nature Milestones: Gene Expression. . (December 2005). doi :10.1038/nrm1798 . http://www.nature.com/milestones/geneexpression/milestones/articles/milegene09.html . ^ “Chromatin structure: a repeating unit of histones and DNA”. Science 184 (4139): 868–71. (May 1974). Bibcode : 1974Sci...184..868K . doi :10.1126/science.184.4139.868 . PMID 4825889 . ^ “Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones”. Cell 49 (2): 203–10. (April 1987). doi :10.1016/0092-8674(87)90561-7 . PMID 3568125 . ^ “Nucleosome loss activates yeast downstream promoters in vivo”. Cell 55 (6): 1137–45. (1988). doi :10.1016/0092-8674(88)90258-9 . PMID 2849508 . ^ “Nucleosome Positioning”. Encyclopedia of Bioinformatics and Computational Biology 2 : 308–317. (2019). doi :10.1016/B978-0-12-809633-8.20242-2 . ISBN 9780128114322 . ^ Alberts, Bruce (2002). Molecular biology of the cell ISBN 978-0-8153-4072-0 . https://www.ncbi.nlm.nih.gov/books/bv.fcgi?highlight=Nucleosome&rid=mboc4.section.608#630 ^ Chromosomal DNA and Its Packaging in the Chromatin Fiber . https://www.ncbi.nlm.nih.gov/books/NBK26834/#:~:text=On%20average%2C%20therefore%2C%20nucleosomes%20repeat,contains%20approximately%2030%20million%20nucleosomes ^ a b “Proximity of H2A.Z containing nucleosome to the transcription start site influences gene expression levels in the mammalian liver and brain” . Nucleic Acids Research 40 (18): 8965–78. (October 2012). doi :10.1093/nar/gks665 . PMC 3467062 . PMID 22821566 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3467062/ .
^ “Nuclear and chromatin composition of mammalian gametes and early embryos”. Biochemistry and Cell Biology 70 (10–11): 856–66. (1992). doi :10.1139/o92-134 . PMID 1297351 . ^ a b c d “Controlling the double helix”. Nature 421 (6921): 448–53. (January 2003). Bibcode : 2003Natur.421..448F . doi :10.1038/nature01411 . PMID 12540921 .
^ Richmond, T. J.; Finch, J. T.; Rushton, B.; Rhodes, D.; Klug, A. (1984 Oct 11-17). “Structure of the nucleosome core particle at 7 A resolution” . Nature 311 (5986): 532–537. doi :10.1038/311532a0 . ISSN 0028-0836 . PMID 6482966 . https://pubmed.ncbi.nlm.nih.gov/6482966 . ^ “Preparative separation of nucleosome core particles containing defined-sequence DNA in multiple translational phases”. Electrophoresis 16 (10): 1861–4. (October 1995). doi :10.1002/elps.11501601305 . PMID 8586054 . ^ “Large-scale production of palindrome DNA fragments”. Analytical Biochemistry 231 (1): 109–14. (1995). doi :10.1006/abio.1995.1509 . PMID 8678288 . ^ “X-ray diffraction analysis of crystals containing twofold symmetric nucleosome core particles”. Acta Crystallographica Section D 52 (Pt 2): 283–8. (1996). doi :10.1107/S0907444995009139 . PMID 15299701 . ^ “Asymmetries in the nucleosome core particle at 2.5 A resolution”. Acta Crystallographica Section D 56 (Pt 12): 1513–34. (2000). doi :10.1107/s0907444900011847 . PMID 11092917 . ^ “Preparation and crystallization of nucleosome core particle”. Methods in Enzymology 375 : 44–62. (2004). doi :10.1016/s0076-6879(03)75003-4 . ISBN 9780121827793 . PMID 14870658 . ^ “Structure and dynamic properties of nucleosome core particles”. FEBS Letters 579 (4): 895–8. (February 2005). doi :10.1016/j.febslet.2004.11.030 . PMID 15680970 . ^ “Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions” . The EMBO Journal 20 (18): 5207–18. (September 2001). doi :10.1093/emboj/20.18.5207 . PMC 125637 . PMID 11566884 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC125637/ . ^ a b Stryer, Lubert (1995). Biochemistry (fourth ed.). New York - Basingstoke: W. H. Freeman and Company. ISBN 978-0716720096
^ a b X-ray structure of the nucleosome core particle at 2.5 A resolution. doi :10.2210/pdb1eqz/pdb . PDB ID: 1EQZ. http://www.rcsb.org/pdb/explore/explore.do?structureId=1EQZ 2012年10月8日閲覧。 .
^ a b “Asymmetries in the nucleosome core particle at 2.5 A resolution”. Acta Crystallographica Section D 56 (Pt 12): 1513–34. (December 2000). doi :10.1107/S0907444900011847 . PMID 11092917 . PDB ID: 1EQZ.
^ Essential cell biology ISBN 0-8153-3480-X . OCLC 52312215 . https://www.worldcat.org/oclc/52312215 ^ “Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 a resolution”. Journal of Molecular Biology 319 (5): 1097–113. (June 2002). doi :10.1016/S0022-2836(02)00386-8 . PMID 12079350 . ^ “A genomic code for nucleosome positioning” . Nature 442 (7104): 772–8. (August 2006). Bibcode : 2006Natur.442..772S . doi :10.1038/nature04979 . PMC 2623244 . PMID 16862119 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2623244/ . ^ “Structures and interactions of the core histone tail domains”. Biopolymers 68 (4): 539–46. (April 2003). doi :10.1002/bip.10303 . PMID 12666178 . ^ “Position and orientation of the globular domain of linker histone H5 on the nucleosome”. Nature 395 (6700): 402–5. (September 1998). Bibcode : 1998Natur.395..402Z . doi :10.1038/26521 . PMID 9759733 . ^ “Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin” . The Journal of Cell Biology 83 (2 Pt 1): 403–27. (November 1979). doi :10.1083/jcb.83.2.403 . PMC 2111545 . PMID 387806 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2111545/ . ^ “X-ray structure of a tetranucleosome and its implications for the chromatin fibre”. Nature 436 (7047): 138–41. (July 2005). Bibcode : 2005Natur.436..138S . doi :10.1038/nature03686 . PMID 16001076 . ^ “EM measurements define the dimensions of the "30-nm" chromatin fiber: evidence for a compact, interdigitated structure” . Proceedings of the National Academy of Sciences of the United States of America 103 (17): 6506–11. (April 2006). Bibcode : 2006PNAS..103.6506R . doi :10.1073/pnas.0601212103 . PMC 1436021 . PMID 16617109 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1436021/ . ^ “Predicting nucleosome positions on the DNA: combining intrinsic sequence preferences and remodeler activities” . Nucleic Acids Research 37 (17): 5641–55. (September 2009). doi :10.1093/nar/gkp610 . PMC 2761276 . PMID 19625488 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2761276/ . ^ “Formation, stability and core histone positioning of nucleosomes reassembled on bent and other nucleosome-derived DNA”. Journal of Molecular Biology 207 (1): 183–92. (May 1989). doi :10.1016/0022-2836(89)90449-X . PMID 2738923 . ^ Van Steensel, Bas, ed (July 2008). “The insulator binding protein CTCF positions 20 nucleosomes around its binding sites across the human genome” . PLOS Genetics 4 (7): e1000138. doi :10.1371/journal.pgen.1000138 . PMC 2453330 . PMID 18654629 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2453330/ . ^ “Rapid spontaneous accessibility of nucleosomal DNA”. Nature Structural & Molecular Biology 12 (1): 46–53. (January 2005). doi :10.1038/nsmb869 . PMID 15580276 . ^ “Nucleosomes facilitate their own invasion”. Nature Structural & Molecular Biology 11 (8): 763–9. (August 2004). doi :10.1038/nsmb801 . PMID 15258568 . ^ “Nucleosomal fluctuations govern the transcription dynamics of RNA polymerase II” . Science 325 (5940): 626–8. (July 2009). Bibcode : 2009Sci...325..626H . doi :10.1126/science.1172926 . PMC 2775800 . PMID 19644123 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2775800/ . ^ “Understanding nucleosome dynamics and their links to gene expression and DNA replication” . Nature Reviews Molecular Cell Biology 18 (9): 548–562. (September 2017). doi :10.1038/nrm.2017.47 . PMC 5831138 . PMID 28537572 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5831138/ . ^ “acetylation and methylation of histones and their possible role in the regulation of RNA synthesis” . Proceedings of the National Academy of Sciences of the United States of America 51 (5): 786–94. (May 1964). Bibcode : 1964PNAS...51..786A . doi :10.1073/pnas.51.5.786 . PMC 300163 . PMID 14172992 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC300163/ . ^ “The language of covalent histone modifications”. Nature 403 (6765): 41–5. (January 2000). Bibcode : 2000Natur.403...41S . doi :10.1038/47412 . PMID 10638745 . ^ “Regulated nucleosome mobility and the histone code”. Nature Structural & Molecular Biology 11 (11): 1037–43. (November 2004). doi :10.1038/nsmb851 . PMID 15523479 . ^ “Histone H4 lysine 91 acetylation a core domain modification associated with chromatin assembly” . Molecular Cell 18 (1): 123–30. (April 2005). doi :10.1016/j.molcel.2005.02.031 . PMC 2855496 . PMID 15808514 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2855496/ . ^ “Charge state of the globular histone core controls stability of the nucleosome” . Biophysical Journal 99 (5): 1577–85. (September 2010). Bibcode : 2010BpJ....99.1577F . doi :10.1016/j.bpj.2010.06.046 . PMC 2931741 . PMID 20816070 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2931741/ . ^ “Nucleosome mobilization catalysed by the yeast SWI/SNF complex”. Nature 400 (6746): 784–7. (August 1999). Bibcode : 1999Natur.400..784W . doi :10.1038/23506 . PMID 10466730 . ^ “SWI/SNF unwraps, slides, and rewraps the nucleosome”. Molecular Cell 11 (2): 391–403. (February 2003). doi :10.1016/S1097-2765(03)00039-X . PMID 12620227 . ^ “Histone H2A/H2B dimer exchange by ATP-dependent chromatin remodeling activities” . Molecular Cell 12 (6): 1599–606. (December 2003). doi :10.1016/S1097-2765(03)00499-4 . PMC 3428624 . PMID 14690611 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3428624/ . ^ “Generation of superhelical torsion by ATP-dependent chromatin remodeling activities”. Cell 103 (7): 1133–42. (December 2000). doi :10.1016/S0092-8674(00)00215-4 . PMID 11163188 . ^ “ATP-driven exchange of histone H2AZ variant catalyzed by SWR1 chromatin remodeling complex” . Science 303 (5656): 343–8. (January 2004). Bibcode : 2004Sci...303..343M . doi :10.1126/science.1090701 . PMID 14645854 . https://zenodo.org/record/1230842 . ^ “Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter”. Cell 115 (6): 751–63. (December 2003). doi :10.1016/S0092-8674(03)00934-6 . PMID 14675539 . ^ “Rapid periodic binding and displacement of the glucocorticoid receptor during chromatin remodeling”. Molecular Cell 14 (2): 163–74. (April 2004). doi :10.1016/S1097-2765(04)00178-9 . PMID 15099516 . ^ “Genome-wide nucleosome positioning during embryonic stem cell development”. Nature Structural & Molecular Biology 19 (11): 1185–92. (November 2012). doi :10.1038/nsmb.2419 . PMID 23085715 . ^ “Translational and rotational settings of H2A.Z nucleosomes across the Saccharomyces cerevisiae genome”. Nature 446 (7135): 572–6. (March 2007). Bibcode : 2007Natur.446..572A . doi :10.1038/nature05632 . PMID 17392789 . ^ “The role of chromatin during transcription”. Cell 128 (4): 707–19. (February 2007). doi :10.1016/j.cell.2007.01.015 . PMID 17320508 . ^ “Chromatin remodelling at promoters suppresses antisense transcription”. Nature 450 (7172): 1031–5. (December 2007). Bibcode : 2007Natur.450.1031W . doi :10.1038/nature06391 . PMID 18075583 . ^ a b “A high-resolution atlas of nucleosome occupancy in yeast”. Nature Genetics 39 (10): 1235–44. (October 2007). doi :10.1038/ng2117 . PMID 17873876 .
^ “Conserved nucleosome positioning defines replication origins” . Genes & Development 24 (8): 748–53. (April 2010). doi :10.1101/gad.1913210 . PMC 2854390 . PMID 20351051 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2854390/ . ^ “Dynamic remodeling of individual nucleosomes across a eukaryotic genome in response to transcriptional perturbation” . PLOS Biology 6 (3): e65. (March 2008). doi :10.1371/journal.pbio.0060065 . PMC 2267817 . PMID 18351804 . http://scivee.tv/node/5532 . ^ “DNA bridging and looping by HMO1 provides a mechanism for stabilizing nucleosome-free chromatin” . Nucleic Acids Research 42 (14): 8996–9004. (August 2014). doi :10.1093/nar/gku635 . PMC 4132745 . PMID 25063301 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4132745/ . ^ “Single-molecule studies of high-mobility group B architectural DNA bending proteins” . Biophysical Reviews 9 (1): 17–40. (February 2017). doi :10.1007/s12551-016-0236-4 . PMC 5331113 . PMID 28303166 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5331113/ . ^ “In vitro reconstitution and analysis of mononucleosomes containing defined DNAs and proteins”. Methods 12 (1): 2–9. (May 1997). doi :10.1006/meth.1997.0441 . PMID 9169189 . ^ “Reconstitution of nucleosome core particles from recombinant histones and DNA”. Methods in Enzymology 375 : 23–44. (2004). doi :10.1016/s0076-6879(03)75002-2 . ISBN 9780121827793 . PMID 14870657 . ^ “Nucleosome positioning on chicken and human globin gene promoters in vitro. Novel mapping techniques”. Journal of Molecular Biology 237 (4): 401–14. (April 1994). doi :10.1006/jmbi.1994.1243 . PMID 8151701 . ^ “Site-Specific Disulfide Crosslinked Nucleosomes with Enhanced Stability” . Journal of Molecular Biology 430 (1): 45–57. (January 2018). doi :10.1016/j.jmb.2017.10.029 . PMC 5757783 . PMID 29113904 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5757783/ . ^ Ferentz, A. E.; Verdine, G. L. (1994). “The Convertible Nucleoside Approach: Structural Engineering of Nucleic Acids by Disulfide Cross-Linking”. In Eckstein, Fritz; Lilley, David M. J.. Nucleic Acids and Molecular Biology . Nucleic Acids and Molecular Biology. 8 . pp. 14–40. doi :10.1007/978-3-642-78666-2_2 . ISBN 978-3-642-78668-6 ^ “Conservative Segregation of Tetrametric Units of H3 and H4 Histones during Nucleosome Replication”. Journal of Biochemistry 170 (1): 15–20. (1990). doi :10.1093/oxfordjournals.jbchem.a122999 . PMID 2332416 . ^ “The p150 and p60 subunits of chromatin assembly factor I: a molecular link between newly synthesized histones and DNA replication”. Cell 81 (7): 1105–14. (June 1995). doi :10.1016/S0092-8674(05)80015-7 . PMID 7600578 . ^ “The RCAF complex mediates chromatin assembly during DNA replication and repair”. Nature 402 (6761): 555–60. (December 1999). Bibcode : 1999Natur.402..555T . doi :10.1038/990147 . PMID 10591219 . ^ “Modifications of H3 and H4 during chromatin replication, nucleosome assembly, and histone exchange”. The Journal of Biological Chemistry 281 (14): 9287–96. (April 2006). doi :10.1074/jbc.M512956200 . PMID 16464854 . ^ “Exchange of histones H1, H2A, and H2B in vivo”. Biochemistry 24 (13): 3080–5. (June 1985). doi :10.1021/bi00334a002 . PMID 4027229 . ^ “Nucleosome assembly protein 1 exchanges histone H2A-H2B dimers and assists nucleosome sliding”. The Journal of Biological Chemistry 280 (3): 1817–25. (January 2005). doi :10.1074/jbc.M411347200 . PMID 15516689 . ^ “ATP-dependent chromatin remodeling shapes the DNA replication landscape” . Nature Structural & Molecular Biology 15 (5): 477–84. (May 2008). doi :10.1038/nsmb.1419 . PMC 2678716 . PMID 18408730 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2678716/ . ^ “Replication-Coupled Nucleosome Assembly and Positioning by ATP-Dependent Chromatin-Remodeling Enzymes” . Cell Reports 15 (4): 715–723. (April 2016). doi :10.1016/J.CELREP.2016.03.059 . PMC 5063657 . PMID 27149855 . https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5063657/ .
小田 琢三「SV40クロマチンの構造と機能 」『生物物理』第20巻第3号、日本生物物理学会 、1980年5月25日、163-173頁、doi :10.2142/biophys.20.163 、2020年7月14日閲覧 。 B. Alberts他 著(中村桂子他 翻訳)『細胞の分子生物学 第5版』ニュートンプレス、2010年。 B. Alberts他 著(中村桂子・松原謙一 監訳)『Essential 細胞生物学 第3版』南江堂、2011年。 平岡泰・原口徳子 編『染色体と細胞核のダイナミクス』化学同人、2013年。 平野達也・胡桃坂仁志 編(実験医学増刊号)『教科書を書き換えろ!染色体の新常識』羊土社、2018年。